|
Dernière mise à jour le
|
|
Informations
|
|
|
Logiciel de visualisation moléculaire en 3D sur PC et Linux. |
|
|
Adresses importantes
|
|
|
Présentation des menus déroulants les plus utilisés
|
|
 |
|
|
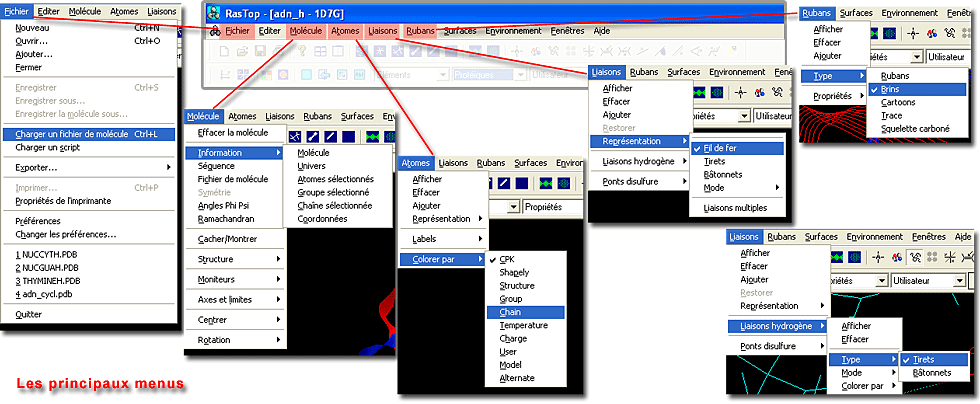
Fichier
|
Molécule
|
Atomes
|
Liaisons
|
Rubans
|
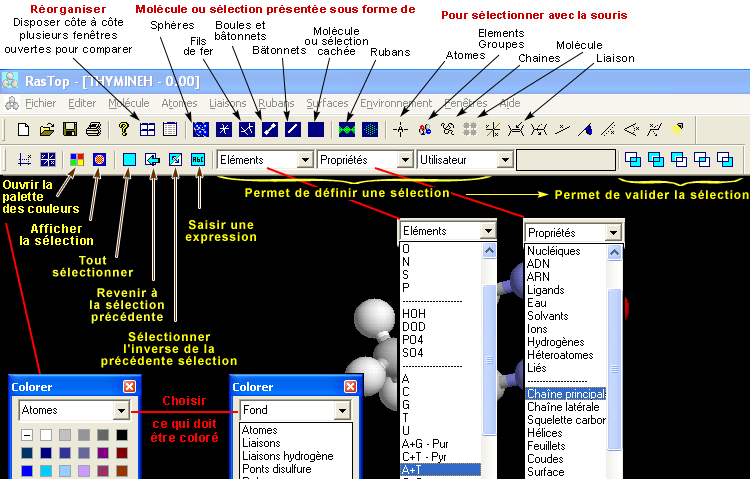
| Nouveau ouvre une nouvelle fenêtre. Ouvrir ouvre un fichier dans une nouvelle fenêtre. Ajouter ajoute une molécule dans une fenêtre contenant déjà une molécule. Charger un fichier de molécule ouvre une molécule dans une fenêtre existante. Exporter permet d'enregistrer la molécule mise en forme sous un format image |
Information Séquence Fichier molécule renseignent sur le nom de la molécule, les chaînes, la liste et le nombre des acides aminés, des atomes, etc. |
Colorer par permet de différencier différentes parties de la molécule (couleurs par défaut modifiables ensuite avec la palette colorer). | Représentation reprend les options disponibles à l'aide des icônes. Liaisons hydrogène permet de les afficher et de choisir le mode de représentation et le couleur (ex. lors de l'affichage d'une molécule d'ADN). Ponts disulfure permet de les afficher et d'en définir le mode de représentation (ex. lors de l'affichage d'un anticorps) |
Afficher reprend la commande à l'aide de l'icône. Type et Propriétés définissent un mode de représentation. |
|
Colorer
|
Menus déroulants de sélection
|
| La palette permet de choisir les couleurs de l'élément sélectionné à l'aide de la souris, via les menus de sélection ou à l'aide du menu déroulant de la palette. Le menu déroulant de la palette permet de choisir les éléments à colorer. Penser par exemple à choisir un fond blanc et à modifier certaines couleurs trop claires, avant d'imprimer. La touche - annule les couleurs et permet de revenir aux couleurs par défaut La touche + ouvre une fenêtre permettant de choisir de nouvelles couleurs. |
Eléments permet de choisir l'élément à sélectionner (un atome, un groupement d'atomes, une portion de la molécule d'ADN, un acide aminé). Propriétés permet de choisir une molécule, une famille de molécule ou une propriété chimique. Valider est indispensable : plusieurs validation sont proposées : nouvelle sélection, ajouter à la sélection, etc. |
|
Déplacement des molécules - Mise en lumière - Informations
|
|
 |
|
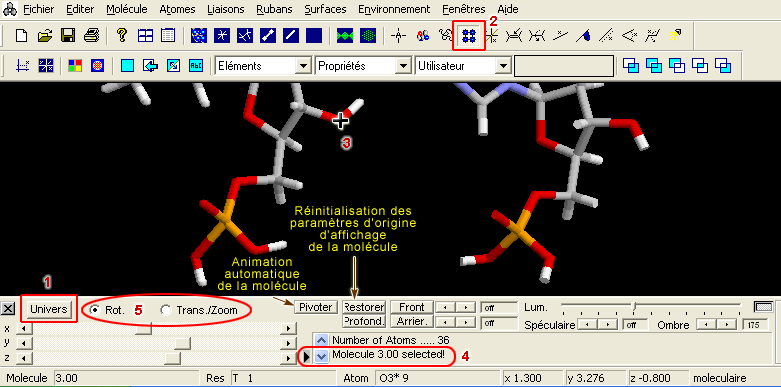
| Manipuler 2 molécules dans une même fenêtre Charger les 2 fichiers dans la même fenêtre (avec la commande Ajouter par exemple)
|
Déplacer la molécule sélectionnée selon les 3 axes (x, y, z)
Les raccourcis clavier indiqués ci-dessous permettent la visite rapide d'une molécule, mais lors des déplacements précis justifiés par une comparaison entre deux molécules, les curseurs s'avèrent être très utiles.
|
| Front permet l'observation de la molécule en son centre, c'est un plan de coupe de la molécule. Avec la flèche de droite, ce plan se déplace en profondeur et efface les atomes de surface. Arrier. a l'effet inverse, il remonte le plan de coupe et efface les atomes profonds. Libérer le bouton réaffiche la molécule entière. Très utile par exemple pour visualiser le contact entre l'enzyme et le substrat, l'anticorps et l'épitope en mode sphères. Lum. - Speculaire - Ombre règlent l'intensité lumineuse et le contraste de la molécule. |
|
| Les petites fenêtres de la ligne inférieure affichent des informations comme le type et le numéro de l'atome sélectionné, ainsi que le nom de la molécule ou de la chaîne, le nom de l'acide aminé et son numéro (pour les protéines), les coordonnées tridimensionnelles. | |
- Autres fiches techniques concernant Rastop sur le Web
| Jean-Paul BERGER Site SVT - Lycée Georges Pompidou - Sharjah - Emirats Arabes Unis |
Si cette page s'ouvre hors cadre Retour à la page d'accueil du site |